|
Monitoreo poblacional de
genotipos de rotavirus en efluentes cloacales y muestras
clínicas
Patricia A. Barril, Miguel O. Giordano, María B. Isa,
Gisela Masachessi, Laura C. Martínez, Silvia V. Nates

Revista Facultad de Ciencias Medicas 2009; 66(Supl.1): 42-47
Laboratorio de Gastroenteritis
Virales y Sarampión. Instituto de Virología “Dr. J. M.
Vanella”, Facultad de Ciencias Médicas, Universidad Nacional
de Córdoba. Enfermera Gordillo Gómez s/n - Ciudad
Universitaria. CP 5000. Córdoba, Argentina.
Introducción
La diarrea aguda constituye un importante problema de salud
pública en los países en desarrollo debido a las altas tasas
de morbi-mortalidad que registra principalmente en niños
menores de 5 años. Según la Organización Mundial de la Salud
se estima que cada año se presentan 1300 millones de
episodios de diarrea en este grupo etareo en los países en
desarrollo y 1.6 millones de muertes por diarrea aguda como
consecuencia de deshidratación, en el 50-70% de los casos.
Los países en vías de desarrollo son el escenario más fértil
para la enfermedad grave ya que desde el punto de vista
ecológico, las diarreas son consecuencia de la infección con
un agente patógeno, más la concurrencia de factores
múltiples de causalidad cuya relación es tan fuerte y
determinante como para considerarlos también factores
etiológicos, siendo los más relevantes las condiciones
sanitarias, el evidente sinergismo con la desnutrición, las
pautas culturales, y el estado inmunitario.
Los virus productores de diarrea se detectaron por primera
vez en la década del ’70 en estudios de heces diarreicas
utilizando el microscopio electrónico y desde entonces la
búsqueda de virus causantes de gastroenteritis ha sido
exhaustiva. En los últimos tres decenios se descubrieron
virus capaces de producir gastroenteritis en niños de corta
edad, entre los que se destacan los rotavirus, astrovirus,
adenovirus y norovirus, en conjunto responsables de
aproximadamente el 60% de los casos de diarrea aguda. Los
estudios de asociación etiológica han demostrado que el
principal agente viral causante de gastroenteritis es el
rotavirus; agente etiológico del 80% de las diarreas
virales. Nuestro grupo de investigación, integrantes del
Laboratorio de Gastroenteritis Virales del Instituto de
Virología “Dr. J. M. Vanella” de la Facultad de Ciencias
Médicas, se encuentra esencialmente abocado al estudio de
este patógeno desde el año 1979. En el camino recorrido se
han realizado aportes al conocimiento de los rotavirus en
las áreas de epidemiología clínica y molecular y diagnóstico
(1,2,3,4).
Los rotavirus poseen un genoma de 11 segmentos de ARN de
doble cadena, rodeado por una triple cápside proteica (5).
Las proteínas que constituyen la cápside externa, VP4 y VP7,
contienen las características antigénicas que inducen en el
huésped una respuesta inmune protectora ante una nueva
exposición al virus. En base a la diversidad de estas
proteínas, los rotavirus grupo A se clasifican en genotipos
P (según la proteína VP4) y G (en base a la proteína VP7).
Los genotipos de mayor prevalencia a nivel mundial y local
asociados con infecciones en humanos son G1, G2, G3, G4, G9,
P[4] y P[8] (6,7).
Desde el año 2006 se encuentran disponibles en el mercado
argentino dos fórmulas vacunales anti-rotavirus, una
monovalente (G1 P[8]) y otra pentavalente (G1-G4 P[8]). Los
estudios de eficacia de ambas vacunas mostraron un alto
nivel de protección contra los genotipos más prevalentes
(8,9). Sin embargo, ante la licencia de toda nueva vacuna es
importante que su introducción sea acompañada de un sistema
integrado de vigilancia epidemiológica clínica y molecular
para monitorear su seguridad e impacto en la disminución de
la enfermedad, así como también la dinámica de circulación
de genotipos en cada región.
En la actualidad, la vigilancia molecular de rotavirus se
basa en el análisis de genotipos virales aislados de casos
clínicos, los cuales sólo incluyen pacientes con
sintomatología lo suficientemente severa como para requerir
atención médica y acota el estudio a uno o unos pocos
centros asistenciales. Para salvar estas limitaciones, sería
importante implementar un método que permita obtener una
visión más global de la epidemiología molecular de los
rotavirus circulantes en la comunidad, incluyendo también en
el análisis molecular los genotipos excretados por
individuos que no demandaron asistencia médica y aquellos
asintomáticos.
El objetivo del presente trabajo fue evaluar si el monitoreo
de las aguas cloacales de la comunidad sería una fuente
alternativa a la de los casos clínicos para investigar la
epidemiología molecular de los rotavirus. En este sentido,
se compararon los genotipos G de rotavirus identificados en
aguas cloacales de la Ciudad de Córdoba con los G tipos
detectados en muestras de pacientes que requirieron atención
médica por diarrea por rotavirus en el Hospital Privado de
la misma ciudad durante el mismo período de tiempo.
Materiales
y
Métodos
Muestras clínicas.
Se analizaron 50 muestras de materia fecal rotavirus
positivas, obtenidas de niños menores de 3 años de edad que
requirieron atención médica en el Hospital Privado de la
Ciudad de Córdoba por un cuadro diarreico agudo. Período de
muestreo: Enero-Diciembre 2006.
Muestras cloacales.
Se colectaron semanalmente muestras de 1.5 L de agua cloacal
del caño colector de la red cloacal que llega como punto
final a la planta de tratamiento de efluentes cloacales Bajo
Grande de la Ciudad de Córdoba (n=52). Esta red cloacal
cubre aproximadamente al 60% de la población de la Ciudad de
Córdoba. Período de muestreo: Enero-Diciembre 2006. Las
muestras fueron concentradas 100X por centrifugación y
precipitación con polietilenglicol (10,11,12)
Extracción de ácidos nucleicos y transcripción
reversa-reacción en cadena de la polimerasa (RT-PCR).
La extracción del ARN viral de las muestras clínicas y
concentrados cloacales se realizó mediante el método de
fenol-cloroformo seguido de precipitación alcohólica de
acuerdo a procedimientos estándares (13). Los ARN extraídos
fueron procesados por RT-PCR con el par de primera
Beg9/End9 para amplificar el gen 9 del genoma de rotavirus
(VP7) (14).
Genotipificación.
Los productos de PCR fueron procesados por heminested-PCR
con un set de primers específicos para los genotipos G1, G2,
G3, G4, G8 y G9, y el primer consenso End9 (14). Los
amplicones obtenidos fueron analizados por electroforesis en
geles de poliacrilamida y los productos revelados por
tinción argéntica (PAGE/SS) (15,16).
Análisis estadístico.
Se utilizó el test Z para comparar proporciones, con el fin
de analizar las frecuencias de detección de G tipos de
rotavirus en muestras clínicas y cloacales (17).
Resultados
Frecuencia de detección de G tipos de rotavirus.
De las 50 muestras clínicas rotavirus positivas colectadas
durante el año 2006, 3 fueron obtenidas durante los meses
cálidos del año (Enero-Marzo) y 47 durante los meses fríos
(Abril-Septiembre). Del total de muestras cloacales
colectadas (n=52), 26 se obtuvieron durante los meses
cálidos y 26 durante los meses fríos; siendo el 100% de las
muestras positivas para rotavirus por RT-PCR seguida de
heminested-PCR. Debido al bajo número de muestras clínicas
rotavirus positivas obtenidas durante los meses cálidos sólo
se realizó la comparación de G tipos detectados por ambos
sistemas de vigilancia analizando los muestreos obtenidos en
los meses fríos del año.
La totalidad de las muestras de materia fecal (n=47) y
cloacales (n=26) fueron genotipificadas y los resultados se
muestran en la Tabla I.
El 31.9% de las muestras clínicas (15/47) correspondieron a
infecciones mixtas de G tipos de rotavirus, alcanzando un
total de 66 G tipos detectados. Del mismo modo, más de un G
tipo fue detectado en el 77% de las muestras cloacales
(20/26), contabilizando un total de 64 G tipos detectados.
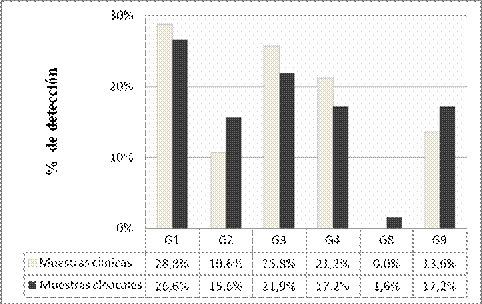
Distribución proporcional de G tipos de rotavirus.
La distribución proporcional de genotipos G de rotavirus
detectados por ambos sistemas de vigilancia durante los
meses fríos se muestra en la
Figura I. Los resultados indican que no existe
diferencia estadísticamente significativa (P>0.05) entre el
análisis de G tipos de rotavirus detectados en muestras
clínicas y cloacales.
Discusión
Está ampliamente descripto que la diarrea por rotavirus es
una patología estacional, característica del período
invernal. Esto se ve reflejado en el presente estudio en el
que el 90% de los muestras de casos clínicos
rotavirus-positivos fueron recolectadas durante los meses
fríos del año. Esto llevaría a concluir que la circulación
del virus estaría acotada al período invernal del año. Sin
embargo, la vigilancia ambiental detectó la circulación de
rotavirus tanto en los meses fríos como en los cálidos,
poniendo en evidencia la circulación continua de rotavirus.
Esto sugeriría que la vigilancia ambiental sería un método
más sensible que la clínica para el monitoreo de los G tipos
virales que circulan en la comunidad. Se desconoce el/las
causa que llevan a un registro significativamente más alto
de diarrea por rotavirus en los meses fríos respecto a los
cálidos. Se postulan factores fisiológicos del hospedador
y/o ambientales que facilitarían el proceso mórbido de la
infección.
La caracterización viral de muestras cloacales mostró un
perfil de circulación de G tipos de rotavirus concordante
con el observado al analizar muestras clínicas. Ambos
sistemas de vigilancia detectaron G1 y G3 como los genotipos
predominantes, seguidos de los tipos G4, G9 y G2. El
genotipo G8 fue sólo detectado en cloacas, siendo este un
genotipo común en ganado bovino, pero atípico en humanos
(excepto en algunos países africanos). Este resultado aporta
una evidencia más de la mayor sensibilidad del muestreo
poblacional respecto a casos clínicos, indicando que el solo
muestreo clínico en un único centro asistencial no
reflejaría en su totalidad los G tipos que circulan en la
comunidad.
Los resultados por otra parte indican que la comparación del
análisis de muestras clínicas y cloacales muestra una imagen
similar de la circulación de los G tipos más frecuentes de
rotavirus en la comunidad (G1, G2, G3 y G4) y que ambos
detectan un aumento en la circulación del genotipo emergente
G9 respecto a las frecuencias anteriormente registradas en
la misma población (7). Este resultado es interesante debido
a que el genotipo G9 no está incorporado en las fórmulas
vacunales disponibles en el mercado y que no hay estudios de
eficacia de inmunidad heterotípica para proteger contra la
infección de cepas rotavirus genotipo G9.
De esta manera, el análisis de muestras cloacales sería un
método sensible para la vigilancia epidemiológica de
genotipos de rotavirus circulantes en una comunidad, que
incorpora los G tipos excretados por todo individuo
rotavirus-infectado, proponiendo un sistema alternativo a la
vigilancia clínica para monitorear la actividad viral y la
diversidad y emergencia de cepas de rotavirus, independiente
del ámbito asistencial.

Bibliografía
1. Giordano,
M O, Ferreyra, L J, Isa, M B, Martinez, L C, Yudowsky, S I,
Nates, S V. the epidemiology of acute viral gastroenteritis
in hospitalized children in Cordoba City, Argentina: an
insight of disease burden. Rev Inst Med Trop S Paulo. 2001;
43:193-198.[Open
Access-Scielo]
2.
Basnec, S N, Giordano, M O, Bennun, F R, Nates, S V,
Vergara, M, Depetris, A R. Detection of two atypical
rotaviruses in the Province of Misiones, Argentina. Acta
Virol. 1991; 35:408-412.
[Abstract]
3.Giordano, M O, Basnec, S N, Nates, S V, Bennun, F R,
Depetris, A R. Rapid techniques for diagnosis and
epidemiological studies of rotavirus infection, J Virol
Methods. 1991; 35:59-63.
[Abstract]
4.Giordano,
M O, Depetris, A R, Nates, SV.
Retrospective study of circulation of human rotavirus
electropherotypes in the city of Cordoba, Argentina,
1979-1989. Rev Arg Microbiol. 1995; 27:1-10.
[Abstract]
5.
Greenberg, H B, Valdesuso, J, van Wyke, K, Midthun,
K, Walsh, M, McAuliffe, V, Wyatt, R G, Kalica, A R, Flores,
J, Hoshino, Y. Production and preliminary characterization
of monoclonal antibodies directed at two surface proteins of
rhesus rotavirus. J Virol; 1983, 47:267-275.
[Fulltext]
6.Widdowson, M A, Bresee, J S, Gentch, J R, Glass, R I.
Rotavirus disease and its prevention. Curr Opin
Gastroenterol; 2004, 21:26-31.
[Full text]
7.Barril, P A, Martinez, L C, Giordano, M O, Castello, A A,
Rota, R P, Isa, M B, Masachessi, G, Ferreyra, L J, Glikmann,
G, Nates, S V. Detection of group A human rotavirus G9
genotype circulating in Córdoba, Argentina, as early as
1980. J Med Virol; 2006, 78:1113-1118.
8.Ruiz-Palacios, G M, Perez-Schael, I, Velazquez, F R,
Abate, H, Breuer, T, Clemens, S C. Safety and efficacy of an
attenuated vaccine against severe rotavirus gastroenteritis.
N Engl J Med; 2006; 354:11-22.
[Full text]
9.Matson, D O. The pentavalent rotavirus vaccine, Rotateq.
Semin Pediatr Infect Dis; 2006, 17:195-199.
[Abstract]
10.Lewis, G D, Metcalf, T G. Polyethilene glycol
precipitation for recovery of pathogenic viruses, including
hepatitis A virus and human rotavirus, from oyster, water,
and sediment samples. Appl Environm Microbiol; 1988,
54:1983-1988.
[Full text]
11.Greening, G E, Hewitt, J, Lewis, G D. Evaluation of
integrated cell culture-PCR (C-PCR) for virological analysis
of environmental samples. J Appl Microbiol; 2002,
93:745-750.
[Full text]
12.Huang, Q S, Greening, G E, Baker, M G, Grimwood, K,
Hewitt, J, Hulston, D,
van Duin, L,
Fitzsimons, A,
Garrett, N,
Graham, D,
Lennon, D,
Shimizu, H,
Miyamura, T,
Pallansch, M A.
Persistence of oral poliovirus vaccine virus after its
removal from the immunization schedule in New Zealand.
Lancet; 2005, 366:394-396.
[Abstract]
13.Perry, R, La Torre, J, Kelley, D, Greemberg, J. On the
lability of poly(A) sequences during extraction of messenger
RNA from polyribosomes. Biochim Biophys Acta; 1972,
262:220-226.
14.Gouvea, V, Glass, R, Woods, P, Taniguchi, K, Clark, H,
Forrester, B, Fang, Z Y. Polymerase chain reaction
amplification and typing of rotavirus nucleic acid from
stool specimens. J Clin Microb; 1990, 28:276-282.
[Full text-PubMed Central]
15.Laemmli U. Cleavage of structural proteins during the
assembly of the head of bacteriophage T4. Nature; 1970,
227:680-685.
[Full text]
16.Herring, A, Inglis, N, Ojeh, C, Snodgrass, D, Menzies, J.
Rapid diagnosis of rotavirus infection by direct detection
of viral nucleic acid silver-stained polyacrylamide gels. J
Clin Microb; 1982, 16:473-477.
[Full text-PubMed Central]
17.Zar, J H. Z-test for comparing two proportions. In:
Biostatistical analysis, 3rd ed. NJ: Electronic
Technical Publishing; 1996.

Figura I.
Distribución proporcional de G tipos de rotavirus detectados
en muestras clínicas (n=66) y cloacales (n=64).
Abril-Septiembre 2006.
Tabla
I.
Número (%) de G tipos de rotavirus detectados en muestras
clínicas (n=47) y cloacales (n=26). Abril-Septiembre 2006.
|
Genotipo G |
Muestras clínicas, n (%) |
Muestras cloacales, n (%) |
|
G1 |
19 (40.4) |
17 (65.4) |
|
G2 |
7 (14.9) |
10 (38.5) |
|
G3 |
17 (36.2) |
14
(53.8) |
|
G4 |
14 (29.8) |
11 (42.3) |
|
G8 |
0 (0) |
1 (3.8) |
|
G9 |
9 (19.1) |
11 (42.3) |

|